Endométrites à Taylorella asinigenitalis
Pourquoi étudier cette bactérie ?
L’espèce Taylorella asinigenitalis, reportée pour la première fois en 1995, est étroitement apparentée à l’agent de la métrite contagieuse équine (Taylorella equigenitalis), maladie de la monte réglementée en France, en Europe et à l’international qui atteint toutes les races équines. La métrite contagieuse équine représente une menace économique pour la filière équine car elle peut fortement perturber la reproduction des équidés (avortements, infertilité transitoire…) et sa circulation est favorisée par la persistance de la bactérie dans l’appareil génital de l’animal, son portage asymptomatique chez l’étalon et l’émergence des échecs thérapeutiques.
A l’inverse, T. asinigenitalis est principalement retrouvée chez les ânes (mais peut également contaminer les chevaux) et est considérée comme « non pathogène » et donc peu étudiée. Néanmoins, elle possède des facteurs de risque de transmission et de persistance similaires à T. equigenitalis, et l’émergence d’un premier foyer naturel d’endométrites sévères à T. asinigenitalis chez 3 juments en 2019 (Emirats Arabes Unis) suggère une virulence potentiellement négligée.
Caractérisation de l’espèce Taylorella asinigenitalis : phylogénie, pathogénicité et résistance aux antibiotiques (projet CaraTasi)
« Le but de ce doctorat est de générer des connaissances académiques, sources de retombées finalisées en médecine vétérinaire, pour une prise en considération de l’émergence d’endométrites aiguës à T. asinigenitalis chez les juments, à l’instar du foyer Emirati de 2019 ».


Sofia KOZAK effectue ses travaux de recherche au sein de l’unité PhEED hébergée dans les locaux de Normandie Équine Vallée à Goustranville.
Dans le cadre de ses travaux de recherche, Sofia KOZAK poursuit un triple objectif :
- Caractériser l’espèce de Taylorella asinigenitalis,
Etudier sa pathogénicité,
Etudier sa résistance vis-à-vis d’un panel d’antibiotiques.
Les perspectives d’application sont les suivantes :
Les outils de génotypage développés permettront une parfaite résolution des liens entre les membres du genre Taylorella afin de cibler les souches les plus à risque.
L’analyse croisée de l’ensemble des données obtenues devrait permettre d’évaluer le caractère pathogène ou non chez le cheval de l’espèce T. asinigenitalis.
Des antibiotiques plus adaptés pourront être proposés pour réduire les échecs de traitement et ainsi diminuer la quantité d’antibiotiques utilisés et la sélection de résistance.
=> Caractériser ≈70 souches de T. asinigenitalis après séquençage de leur génome, associé à la mise au point d’une core genome MLST commune au genre Taylorella.
Cette approche complémentera la méthode de génotypage actuelle par MLST. Les SNP seront également mis en évidence par rapport au génome de référence déjà publié.

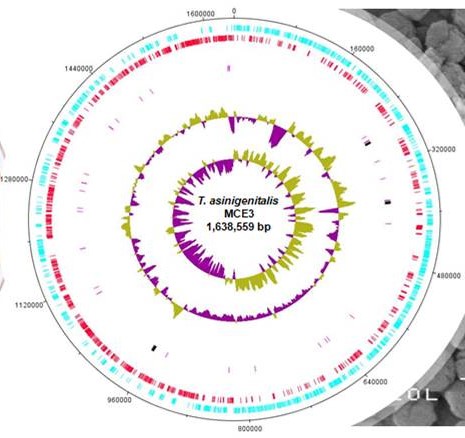
Taylorella asinigenitalis à l’échelle chromosomique : cette image représente la « carte » du chromosome de T. asinigenitalis, le chromosome chez cette bactérie étant circulaire et en un seul exemplaire.
Les cercles bleu et rouge représentent les zones du chromosome qui sont susceptibles d’être traduites en protéines (des séquences codantes) lues dans le sens horaire (bleu) et le sens anti-horaire (rouge).
Plus on se rapproche du cœur de la figure, plus les informations données sur la structure génétique sont précises (en rose les ARN de transfert, en noir les ARN ribosomiques puis en violet et olive le GC%).
==> Etudier la pathogénicité de T. asinigenitalis par des essais d’invasion et de réplication en cellules de derme équin (ED) combinés aux données génomiques obtenues.
Des observations en microscopie électronique seront également réalisées pour visualiser une possible localisation intracellulaire.
===> Etudier la résistance de T. asinigenitalis vis-à-vis d’un panel défini d’antibiotiques et comparer les résultats avec ceux de T. equigenitalis.
La résistance à ce panel d’antibiotiques sera ensuite étudiée en co-culture avec des cellules ED afin de prendre en compte la possible localisation intracellulaire des Taylorella sur la résistance aux antibiotiques testés. En fonction des résultats de résistance aux antibiotiques, et entre autres des résultats de l’évaluation de l’impact du caractère intracellulaire du genre Taylorella dans la résistance aux antibiotiques usuels pour la MCE, un ou plusieurs antibiotiques plus appropriés pourront être proposés aux vétérinaires traitants afin de réduire/éliminer les échecs de traitement, et finalement diminuer la quantité d’antibiotiques utilisés et donc les coûts de traitement et le risque de sélection de bactéries résistantes.

Taylorella asinigenitalis à l’échelle macroscopique : cette image représente un antibiogramme, une technique qui permet d’étudier la susceptibilité des souches bactériennes vis-à-vis de différents antibiotiques. Pour cela, une solution contenant la souche à étudier est ensemencée sur une gélose (ici une gélose couleur “chocolat”, qui est en fait composée de sang de cheval cuit), puis des disques cartonnées imbibés d’antibiotique sont déposés à la surface de la gélose.
Après plusieurs jours d’incubation, un tapis bactérien se forme et des halos peuvent être visibles ou non.
La présence de halos indique les zones où la croissance de la bactérie a été inhibée par l’antibiotique (si aucun halo n’est visible autour d’un disque, cela signifie que l’antibiotique n’a aucun effet sur la souche). La taille des halos est mesurée et, selon des règles de lectures bien définies, on peut les interpréter afin de catégoriser la souche de « sensible » ou « résistante » (ou « intermédiaire ») à l’antibiotique.
Relationship between rifampicin resistance and RpoB substitutions of Rhodococcus equi strains isolated in France
