
2022-2025
#bactéries #virus
Projet AVORT-EQ

Coordination scientifique :
Dr. Albertine LEON-SECK (PhD, HDR)

Équipe projet au sein de LABÉO :
Clément PILLON (Doctorant), Elena KOKABI (Ass. Rech. & Dév.)
Unité de recherche d’appartenance (UNICAEN) : DYNAMICURE (UMR 1311 INSERM)

Partenaires :
RESPE pour leur expertise en épidémiologie, analyse de donnée et réseau de vétérinaires sentinelles ; Anses LSAn, Site de Normandie pour les autopsies, mise à disposition de la base de données RESUMEQ ; Prof. J-F Bruyas (ONIRIS) dans la réalisation des prélèvements vaginaux.
Financeurs :

Dans la filière équine, l’avortement est considéré comme un échec majeur pouvant conduire à des pertes économiques importantes. En effet, tout l’effectif de juments gestantes d’un haras peut être touché à cause du caractère contagieux du pathogène incriminé. C’est pourquoi, il est important d’identifier rapidement la nature de l’agent abortif afin de mettre en place les mesures de contrôle adaptées.
Malgré l’évolution des connaissances sur les pathogènes abortifs, 7 à 40% des cas d’avortements infectieux restent inexpliqués selon les régions du monde. En France, le dernier bilan date de 2011 et porte sur 1 822 avortons autopsiés entre 1986 et 2009 par le Laboratoire de Santé Animale, site de Normandie (LSAn), de l’Anses. Dans cette étude, la cause de l’avortement a pu être déterminée uniquement dans 75% des cas, avec la répartition suivante : cause infectieuse 48%, cause non infectieuse 27% et cause indéterminée 25%. Parmi les causes infectieuses, 80% étaient d’origine bactérienne, 15% d’origine virale et 2% d’origine fongique.
Un autre travail de recherche, mené entre 2002 et 2006, dans le cadre de la thèse de doctorat d’A. LEON (dirigée par LABÉO en partenariat avec le LSAn) avait conduit à l’exploration étiologique de 407 cas d’avortement dont 53% étaient d’origine infectieuse. Pour cette étude, 17,5% des cas sont restés sans explication et parmi les 216 avortements infectieux, l’agent étiologique n’a été identifié que dans 105 cas (48,6%). Ce projet a permis de développer des outils de détection pour les herpès virus équins de type 1 et 4 (HVE-1 et 4), le virus de l’Artérite Virale Equine (AVE) ainsi que pour les leptospires. Il a conduit à la création d’un sous-réseau avortement au sein du Réseau d’Épidémiosurveillance en Pathologie Équine (RESPE) fin 2008.
Depuis lors, LABÉO, dans le cadre de son activité de soutien à la filière équine, réalise par PCR les analyses de première intention citées ci-dessus. LABÉO a continué à développer son expertise en mettant au point des tests PCR ciblant 1) des pathogènes (tels que Coxiella burnetii ou Neospora caninum) occasionnellement rapportés, ou suspectés, 2) des agents abortifs majeurs chez d’autres espèces telles que les bovins ou l’Homme. Ce panel de tests constitue les analyses de seconde intention.

Clément PILLON effectue ses travaux de recherche dans les locaux de Normandie Équine Vallée à Saint-Contest.
Commencé en janvier 2022, ce projet de thèse comprend trois actions interconnectées :
Réaliser une étude rétrospective descriptive des avortements et de leur diagnostic dans les laboratoires d’analyse français depuis 2008, afin de proposer une optimisation de la surveillance des avortements au niveau national. Pour cela, les bases de données disponibles (RESPE et RESUMEQ notamment) seront investiguées afin de définir les indicateurs épidémiologiques les plus pertinents à suivre selon la méthode de capture/recapture.
Évaluer les technologies innovantes de séquençage haut débit (NGS) pour l’exploration des étiologies non résolues et les confronter aux résultats d’un diagnostic ciblé. Les résultats obtenus (autopsie, histologie, culture, bio. mol., NGS) sur un panel d’avortons seront comparés et permettront de formuler des hypothèses quant à la présence de « nouveaux » agents étiologiques abortifs.
Mettre au point une méthode prédictive du risque d’avortement infectieux en se basant sur l’analyse par NGS de prélèvements vaginaux qui devra permettre d’anticiper, voire de limiter/réduire le risque d’infection en cours de gestation.
L’action 1) a permis d’inclure 203 chevaux répartis en trois groupes : M (n=38), S (n=83) et PAs (n=82). Un article à comité de lecture international a été soumis. Il présentait les résultats comparatifs d’ELISA, PCR, de culture sur ENP vs LPG sur les chevaux du groupe malade. Les reviewers ont demandé d’inclure tous les chevaux de l’étude dans cette analyse. Une nouvelle analyse des données a été réalisée et l’article est en cours de mise à jour. Au total, 55 souches ont été isolées. Leur caractérisation phénotypique n’a pas montré de résistance aux antibiotiques. La caractérisation moléculaire par NGS a été réalisée sur 18 souches. Elles ont été caractérisées comme appartenant à la séquence type (ST 79) par MLST, considérée comme une souche fréquemment retrouvée en Europe. Des analyses plus approfondies sur les gènes de virulence, résistance, persistance ou d’échappement du système immunitaire sont en cours, en collaboration avec les Drs Wilson et Waller. Le séquençage complet des autres souches sera réalisé à LABÉO en 2023.
Les biofilms de neuf souches isolées au cours de cette étude [trois S. equi isolées de chevaux M, trois de chevaux PAs et trois S. equi subsp zooepidemicus (S. zoo), bactéries connues pour former du biofilm] ont été caractérisés et comparés. Quatre technologies ont été utilisées : trois en statique (la coloration au cristal violet, l’impédancemétrie et la microscopie 3D à fluorescence) et une en dynamique (la microfluidique). Après une phase d’optimisation, chaque technologie a été effectuée en triplicat. Les résultats obtenus ne montrent pas de variation de la production de biofilm selon le type de portage des chevaux (M versus PAs), mais plutôt en fonction de la morphologie de la colonie des souches. En effet, les trois souches de S. equi formant du biofilm présentent de petites colonies en milieu gélosé et sédimentent en milieu de culture liquide. Le même phénotype est observé pour les trois souches de S. zoo. Les trois souches de S. equi peu ou non-productrices de biofilm, présentent quant à elles de grosses colonies sur milieu gélosé et poussent de manière homogène en milieu liquide. Ces résultats sont en cours de valorisation par un second article. Sans candidat, l’action 3 n’a pas pu être réalisée.
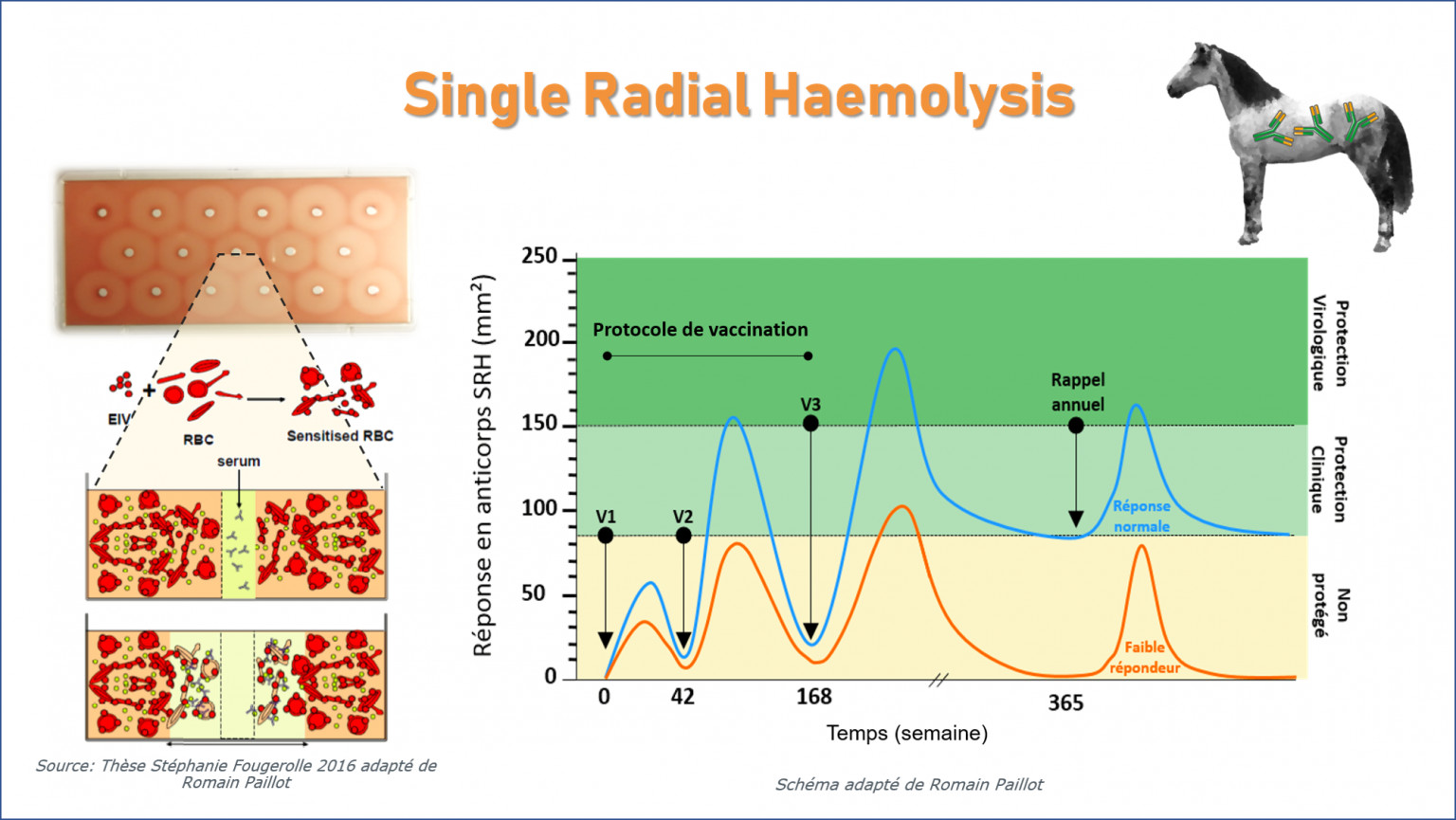
Single Radial Haemolysis (SRH) antibody titres
Paillot R, Sutton G, Thieulent C, Marcillaud-Pitel C, Pronost S.
New EHV-1 variant identified
Vet Rec. 2020 May 30;186(17):573.
Étude de la production de biofilm, de la résistance aux antibiotiques et aux biocides chez des souches de Pseudomonas aeruginosa isolées chez l’Homme et l’animal
MICROBES – 15ème congrès national de la Société Française de Microbiologie, Sep 2019, Paris, France
Decreased susceptibility to didecyldimethylammonium chloride among clinical strains of Pseudomonas aeruginosa, a ten years retrospective study, at the University Hospital of Caen, France
The 31st European Congress of Clinical Microbiology & Infectious Diseases (ECCMID), Jul 2021, online, Austria.
Le projet AVORT-EQ est donc un projet collaboratif, construit avec le RESPE, l’Anses LSAn (site de Normandie), ONIRIS et LABÉO tout en s’appuyant sur l’expertise technologique de l’équipe DYNAMICURE.


