Herpèsvirus équin 1 (EHV-1)

Pourquoi étudier ce virus ?
L’herpèsvirus équin 1 (EHV-1), classé parmi les Herpesviridae, est l’un des pathogènes les plus fréquemment rencontrés au sein de la population équine. Il est aussi couramment appelé virus de la rhinopneumonie.
Il peut être à l’origine de formes de maladie respiratoire, abortive ou néonatale, oculaire ou nerveuse pouvant conduire à l’euthanasie de l’animal. Les mesures de prévention incluant la vaccination constituent la première barrière dans la lutte contre ce virus. En cas d’épizootie, le suivi et l’identification des souches peuvent constituer une aide précieuse pour la compréhension des modes d’infection.
Étude phylogénique de souches d’alphaherpèsvirus et développement d’un outil innovant pour la mesure des anticorps neutralisants

Gabrielle SUTTON a effectué ses travaux de recherche au sein de l’unité BIOTARGEN de l’Université de Caen Normandie hébergée dans les locaux de Normandie Équine Vallée à Saint-Contest.
Le typage de 137 souches d’EHV-1 pour le polymorphisme en position 2254 du gène de l’ADN polymérase (ORF30) associé à la pathogénicité des souches a montré une association significative des souches de type A2254 avec les cas d’avortements.
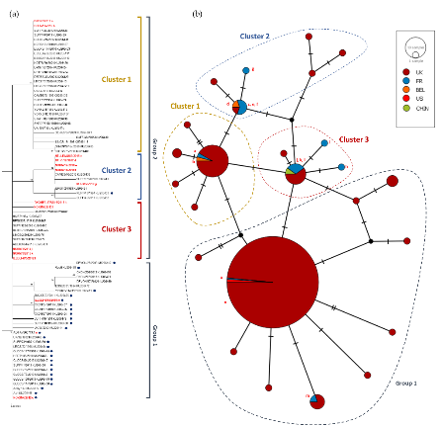
Quatorze souches d’intérêt ont ensuite été analysées par séquençage de l’ORF30 et Multi Locus Sequence Typing (MLST). Le séquençage complet de l’ORF30 s’est montré peu discriminant mais a permis de mettre en évidence un troisième génotype (C2254 (H752)) en position 2254 de l’ORF30. L’analyse par MLST a permis de classer ces souches parmi 7 des 10 clades UL décrits par l’outil à ce jour. Deux autres souches d’herpèsvirus d’équidés, une d’EHV-8 et une d’EHV-9, ont pu être mises en évidence grâce au séquençage de l’ORF30.
Ces différentes souches d’alphaherpèsvirus des équidés ont été caractérisées, comparées et leur sensibilité vis-à-vis de sept molécules antivirales a été évaluée in vitro. Leur proximité génétique a pu être visualisée par la construction d’un network phylogénétique basé sur les séquences de l’ORF30.
Une seconde partie du travail a porté sur le suivi du niveau de protection conféré par les anticorps neutralisant l’EHV-1. Une technique innovante de quantification a été mise au point grâce à la technologie xCELLigence® sur cellules E. Derm et a permis de déterminer les titres induisant 50% de la neutralisation d’une concentration de virus (NT50) à partir d’une population d’échantillons qualifiés provenant d’une infection expérimentale avec la souche d’EHV-1 C2254. Les valeurs de NT50 obtenues ont montré une bonne corrélation avec les valeurs de titres obtenues avec la méthode de référence sur cellules RK13. Les réponses de chevaux à la vaccination ont ensuite pu être étudiées selon le protocole de vaccination reçu et selon l’historique des chevaux.
Paillot R, Sutton G, Thieulent C, Marcillaud-Pitel C, Pronost S.
New EHV-1 variant identified
Vet Rec. 2020 May 30;186(17):573.
